Cada vez está más claro que el SARS-CoV-2 ha venido para quedarse con nosotros como un quinto coronavirus que irá discurriendo por el mundo produciendo catarros más o menos estacionales.
Se unirá a los cuatro coronavirus que, invierno tras invierno, nos provocan resfriados. A saber: dos alfacoronavirus (HCov-229E y HCov-NL63) y dos betacoronavirus (HCov-OC43 y HCoV-HKU1). Su momento álgido en cuanto a infecciones coincide en el tiempo (y en síntomas) con otros virus respiratorios como los rinovirus, los adenovirus y los ortomixovirus (los de la gripe). Y eso hace que nos cueste distinguirlos.
Es lógico plantearse si esos coronavirus se comportaron siempre así. ¿Cómo llegaron a convertirse en virus endémicos humanos? ¿Produjeron pandemias como la actual en algún momento?
Parece que la respuesta a esta última pregunta es sí. Es más, en alguno de los casos ni siquiera hace demasiado tiempo. Todo apunta a que el HCoV-OC43, por ejemplo, se unió al plantel de virus catarrales humanos hace unos 130 años y el HCoV-NL63 entre los siglos 13 y 15.
La gripe rusa de 1890
Actualmente disponemos de conocimientos y herramientas moleculares que nos permiten aislar y analizar los microorganismos que producen enfermedades contagiosas. Sin embargo, hubo una época en la que se desconocía la existencia de estos microorganismos y no se disponía más que del análisis de los síntomas. En aquel momento, casi todas las infecciones respiratorias eran diagnosticadas como gripe.
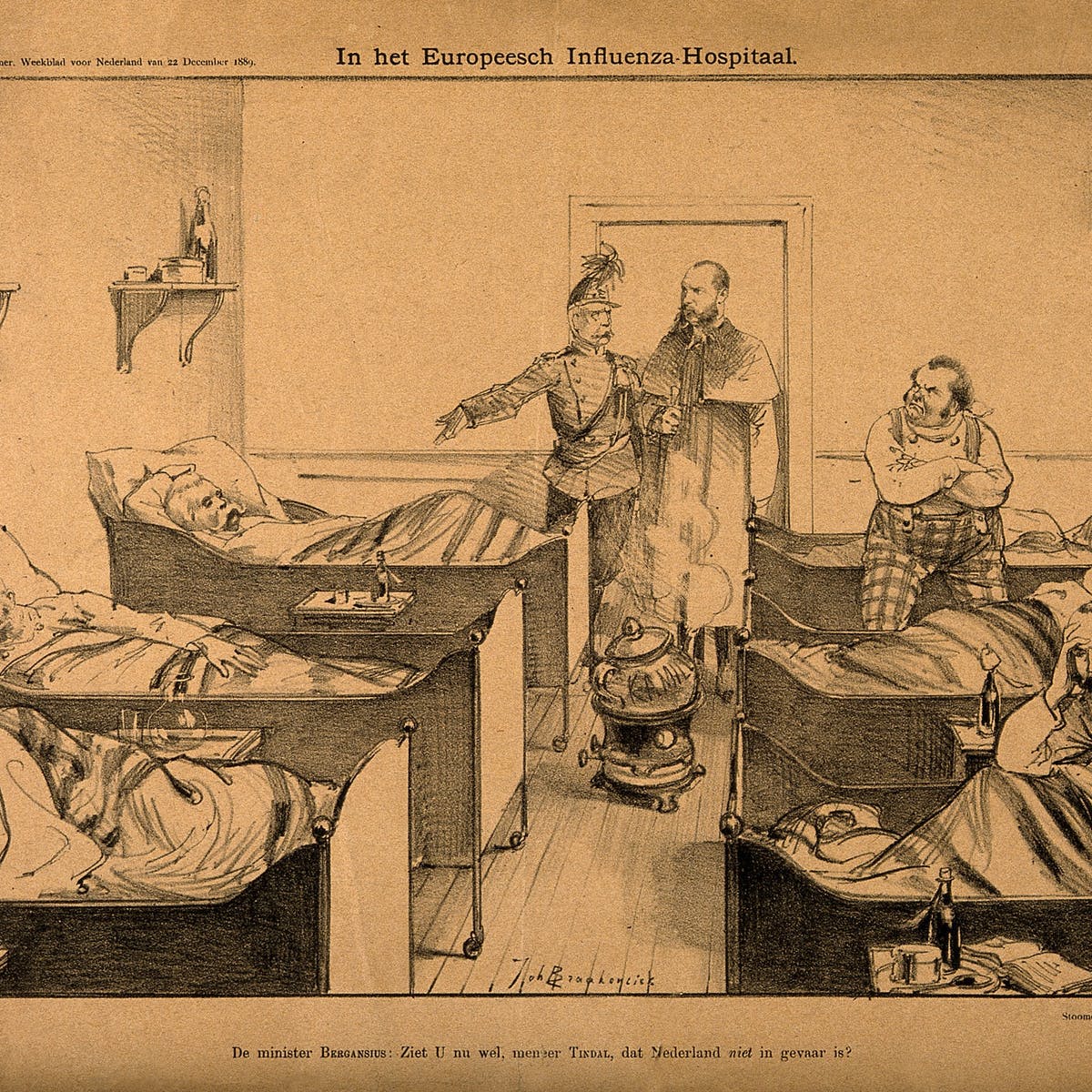
A través de los anales de la época y de un puñado de publicaciones científicas sí que conocemos que en 1889 se reportó un brote de gripe en San Petersburgo que en poco tiempo acabó afectando a toda Europa y Norteamérica. Por eso esta pandemia es conocida como la gripe rusa de 1890.
En un estudio de 2018 realizado sobre la incidencia de esta pandemia en Madrid se constató que los síntomas presentaban un perfil similar a la pandemia de SARS-CoV-2 actual. La población infantil apenas fue afectada y se produjo un aumento progresivo de la mortandad con la edad. Además, hubo más bajas entre la población mayor de 70.
En otro trabajo publicado en 1891, H. Franklin Parsons describió pormenorizadamente los diferentes casos reportados de personas afectadas por esta pandemia. En este estudio se resaltan las particularidades de esta extraña gripe que incluían neumonía, tos seca, dolor de cabeza o diarrea entre otras. Estos síntomas no eran ni son comunes en la gripe habitual, pero sí totalmente compatibles con el contagio con HCoV-OC43 y con el de SARS-CoV-2.
No es descartable que el proceso de transmisión del virus que generó la pandemia de 1890 fuese más lento de lo que se suponía. En esos años, entre 1885 y 1890, hubo una serie de casos extraños en los que consumidores de leche sufrían síntomas como dolor de garganta. Por otro lado, en Oceanía se produjeron varios brotes de gripe cuyos síntomas se confundían o no coincidían con la gripe común.
La falta de más datos clínicos y la imposibilidad de acceder al microorganismo causante de aquella pandemia hace difícil llegar a conclusiones claras, aunque un reciente estudio ha profundizado en las patologías descritas por la literatura científica de la época encontrando grandes similitudes con la infección por coronavirus y menos con la patología de la gripe.
Wikimedia Commons / Wellcome Collection, CC BY
Pistas moleculares de las antiguas pandemias
La tecnología actual nos permite en muy poco tiempo aislar y secuenciar los patógenos que nos afectan. En los últimos años, las técnicas de secuenciación del genoma han permitido no solo diagnosticar rápidamente sino establecer relaciones evolutivas entre los patógenos que nos afectan. Gracias a ello se identificó a toda velocidad el origen del SARS-CoV-2, aunque aún queden lagunas en cuanto a su evolución hasta nosotros.
Sin embargo, los datos de anteriores pandemias son más complejos de seguir. Por lo pronto, la disponibilidad del patógeno que originó la pandemia es prácticamente nula, a no ser que se pueda obtener a partir de exhumaciones de fallecidos sospechosos de haber sufrido la infección.
Para seguirle la pista al origen de los coronavirus humanos que producen catarros comunes no queda otra que echar mano de estudios evolutivos a partir de genomas de virus relacionados y de las cepas actuales.
Hace unos meses, el profesor Marc Van Ranst, experto en coronavirus de la Universidad de Leuven, en Bélgica, indicó que los estudios evolutivos del HCoV-OC43 y de su posible origen, el coronavirus que afecta al ganado bovino, el BCoV-ENT, indican una separación evolutiva de unos 130 años aproximadamente. Es decir, justo cuando se produjo la pandemia de gripe rusa.
El HCoV-OC43 presenta altísima homología con la secuencia génica del BCoV-ENT y también con la del coronavirus de rata HKU24 por lo que no se descarta una posible transmisión de los roedores al ganado bovino y de éste a los humanos.
Por otro lado, es interesante constatar que las secuelas de la COVID-19 son muy parecidas a las secuelas referenciadas para la pandemia de gripe de 1890 y para la de 1918. Estas secuelas incluyen la “niebla mental” y la fatiga crónica, posiblemente indicando un efecto secundario de la inflamación crónica asociada con las infecciones causadas por estos virus y que es común tanto a coronavirus como a ortomixovirus.
Todo ello hace pensar que la pandemia de 1890 no fue causada por un virus de la gripe sino por un coronavirus. Este se habría quedado con nosotros en la forma del HCoV-OC43.
Diferencias entre la pandemia actual y las previas
Entre las pandemias pretéritas y la actual existen diferencias notables. La demografía y la capacidad de movilidad han aumentado enormemente la capacidad de diseminación de los patógenos, especialmente de los respiratorios.
Debemos entender que los patógenos requieren de vectores y de congregación de hospedadores. Los microorganismos similares a los causantes de las pandemias pasadas se dispersan más rápidamente y de manera más global en la actualidad, tal y como ya hemos podido comprobar.
Hoy en día disponemos de herramientas moleculares muy poderosas capaces de aislar e identificar patógenos en pocas semanas. Eso nos permite diseñar terapias más efectivas y, lo que es más importante, diseñar la terapia preventiva más precisa y potente jamás desarrollada: la vacuna.
Afortunadamente, el conocimiento de la diversidad de estos microorganismos y el análisis molecular de sus componentes nos permitirán diseñar mejores y más efectivas vacunas en un futuro no muy lejano.![]()
Guillermo López Lluch, Catedrático del área de Biología Celular. Investigador asociado del Centro Andaluz de Biología del Desarrollo. Investigador en metabolismo, envejecimiento y sistemas inmunológicos y antioxidantes., Universidad Pablo de Olavide
Este artículo fue publicado originalmente en The Conversation. Lea el original.
El periodismo independiente necesita del apoyo de sus lectores para continuar y garantizar que las noticias incómodas que no quieren que leas, sigan estando a tu alcance. ¡Hoy, con tu apoyo, seguiremos trabajando arduamente por un periodismo libre de censuras!
Apoya a El Nacional







